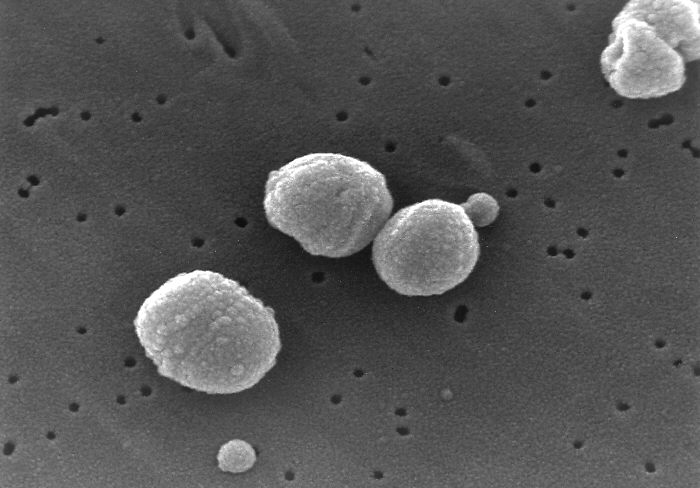
La bacteria Gram positiva Streptococcus pneumoniae al microscopio electrónico
Streptococcus pneumoniae es uno de los patógenos más fastidiosos con los que tiene que enfrentarse la humanidad. Su nombre de pila es neumococo y nos dice que es uno de los muchos microorganismos que producen neumonía. Pero su infección también puede causar otras enfermedades, sobre todo en niños y ancianos. Entre ellas están algunas como sinusitis, otitis media, bacteriemia o incluso meningitis.
Actualmente el neumococo es el causante de 1,6 millones de muertes en todo el mundo cada año. Se está intentando desarrollar vacunas, pero la cosa no es nada fácil. El motivo es que el neumococo está rodeado de una cápsula polisacarídica. Esta cápsula es como una armadura que defiende al neumococo de los ataques de las células de nuestro sistema inmune. Normalmente, las vacunas se diseñan contra los componentes de esas cápsulas. Así nuestro sistema inmune reconoce mejor sus puntos débiles y puede destruir a la bacteria. Lo malo es que el fondo de armario del neumococo no tiene nada que envidiar al de nuestra actual vicepresidenta. Se han descrito 91 tipos distintos de cápsulas, por lo que nos podemos hacer una idea de lo difícil que es diseñar una vacuna que sea efectiva contra esas 91 armaduras.
Otra forma de combatir al neumococo es mediante el uso de antibióticos. La aparición de estas sustancias en la segunda mitad del siglo pasado permitió una lucha efectiva contra las infecciones causadas por dicha bacteria. Desgraciadamente, el uso y abuso de los antibióticos ha producido la aparición de un gran número de cepas resistentes a los mismos. Es por ello que se está buscando con ahínco nuevas moléculas con potencial antibacteriano y para las que no sea fácil la aparición de resistencias.
Recientemente el equipo liderado por el profesor Jesús Sanz de la Universidad Miguel Hernández en colaboración con otros grupos europeos ha diseñado un tipo de moléculas que puede abrir un nuevo camino en la lucha contra el pneumococo. Y lo mejor es que precisamente no actúan sobre la armadura del neumococo, sino sobre lo que está debajo de ella.
Como se ha indicado más arriba, el neumococo es una bacteria Gram postiva. Eso quiere decir que la envoltura de la bacteria tiene la estructura siguiente: una membrana plasmática rodea al citoplasma (banda azul). A su vez la membrana es rodeada por una pared celular compuesta de peptidoglicano y ácidos teicoicos. Aunque el modelo biofísico de la pared de las bacterias aún está siendo dilucidado, podemos imaginarnos que una bacteria es como un edificio en el que la pared está hecha como si fuera hormigón armado y en ese caso el peptidoglicano es como una especie de cemento elástico y los ácidos teicoicos son como las varillas de acero que lo atraviesan y le dan su fortaleza. La cápsula de la que hemos hablado antes serían las placas de mármol o de aluminio que ponemos por fuera para embellecer al edificio.
En la parte de arriba se muestra un esquema de la envoltura del pneumococo. En azul oscuro se representa la membrana citoplasmática. Los hexágonos enlazados de azul claro sería el peptidoglicano. Las líneas verticales rojas y marrones segmentadas son los ácidos teicoicos y en verde se muestran la fosfocolina, lugar de unión de las CBP. En la parte de abajo se muestra la estructura del dominio de unión a colina de la amidasa LytA, la CBP mejor conocida (origen de la imagen).
El caso es que los ácidos teicoicos del neumococo están decorados con grupos de fosfocolina. Esos grupos le sirven a la bacteria para que se peguen una serie de Proteínas de Unión para Colina o CBP (Coline Binding Protein) que deben de realizar su función en el exterior de la célula. Siguiendo con la analogía del edificio, imaginemos que se necesita que unos operarios realicen tareas de mantenimiento externo de la fachada y para ello necesitan asegurarse a una serie de ganchos que se disponen en las barras de acero del hormigón armado. La fosfocolina serían esos ganchos. Evidentemente los operarios llevarían un mecanismo de anclaje a esos ganchos o bolsillos de unión a colina. Entre esas proteínas que cumplen su función en el exterior de la célula hay algunas implicadas en la finalización de la división celular, otras en la producción de toxinas y otras involucradas en la adherencia al hospedador.
La idea desarrollada por el grupo de Jesús Sanz fue la siguiente. Si evitamos que esas proteínas CBP se unan a la fosfocolina de la pared celular no podrán cumplir su función y por lo tanto la bacteria no podrá dividirse, no podrá liberar toxinas y no podrá adherirse al hospedador. Una forma sencilla de comprobar dicha hipótesis es tomar un cultivo de neumococo y añadirle una gran cantidad de colina. Cuando se hace esto, las CBPs se unen a la colina que se ha añadido y no a la fosfocolina de la pared celular. Entonces las células no pueden separarse y forman largas cadenas y además dejan de producir toxinas.
Pero el problema es que la colina no puede ser usada terapéuticamente, al menos en las cantidades requeridas para producir dichos efectos si se lo administraramos a un paciente. Las CBPs tienen más afinidad por las fosfocolinas debido a que se presentan agrupadas en los ácidos teicoicos. Para resolver el problema los investigadores han diseñado un dendrímero, una molécula polimérica ramificada con forma de árbol, y que presenta una gran cantidad de sitios análogos a la colina en sus ramas y a los que las CBPs se unen. La estructura resultante mimetiza a la de los agrupamientos de fosfocolina de los ácidos teicocicos. Y las CBP's se unen con bastante afinidad a ellos. Tanto que dejan de cumplir su función para la célula. Y las concentraciones de uso son tan pequeñas que pueden ser utilizados como agentes terapéuticos
Estructura de los diferentes dendrímeros de poli(propileno imina) usados. Cada punto negro al final de las ramas es un grupo de colina (origen de la imagen) .
Pero lo mejor de la estrategia de usar estos dendrímeros como agentes antibacterianos es que es muy difícil que las bacterias puedan desarrollar resistencias frente a los mismos. La colina es parte del ácido teicoico y por ello es usada por muchas CBPs, luego la bacteria no puede sustituirla por otra cosa así como así. Es como si en los edificios tuvieran que cambiar los barrotes de acero del hormigón armado por barrotes de otro material. Tampoco pueden cambiar los dominios de unión a colina que se encuentran en las CBPs, porque en ese caso ya no se unirían a la fosfocolina de la pared y tampoco podrían cumplir su función. Los dendrímeros están engañando a las CBP's.

Efecto de la colina y de los dendrímeros en cultivos de neumococo. En el control se observan células normales y a su derecha el efecto de la colina. En las demás se indica el nombre de los diferentes dendrímeros mostrados en la figura anterior. Estos se usaron a concentraciones de 100 micromolar. En comparación la colina se uso a una concentración 500 veces superior (origen de la imagen).
Audio 1 y 2 en "El podcast del microbio"